DOP-PCR法检测单抗残余DNA的探讨
DOP-PCR法检测单抗残余DNA的探讨
李萌,武刚,于传飞,王兰*
(中国食品药品检定研究院,卫生部生物技术产品检定与标准化重点实验室,北京100050)
摘 要 目的:利用简并引物(Degenerate Oligonucleotide-Primed,DOP)PCR法,建立一种物种非特异性的宿主细胞残余DNA检测方法。方法:设计简并引物,优化实时定量PCR的条件,从而建立DOP-PCR法,检测3种重组单抗药物宿主细胞表达系统(CHO细胞、SP2/0细胞和NS0细胞)的残余DNA。结果:简并引物选择5`Tag-N6-ATGTGG 3`,定量PCR条件优化为:低严谨性扩增的退火温度由32℃以3%的斜率升至72℃,严谨性扩增后的荧光检测条件优化为77℃,30s。3个物种的DNA扩增曲线R2均能达到0.99以上,回收率达到80%~132%,3次测定的RSD 均小于25%。同时DOP-PCR与CHO细胞特异性的定量PCR进行比较,二者回收率的均值均在80%~130%之间,3次测定的RSD 均小于20%。结论:初步建立了物种非特异性的宿主细胞残余DNA的DOP-PCR检测方法,为单抗药物宿主细胞残余DNA检测提供新思路。
关键词 简并引物,实时定量PCR,残余DNA,单抗药物
中图分类号 Q789
Discussion of Detecting Residual DNA of Monoclonal Antibody by DOP-PCR
Li Meng, Wu Gang, Yu Chuanfei, Wang Lan*
(National Institutes for food and drug Control, Key Laboratory of the Ministry of Health for Research on Quality and Standardization of Biotech Products, Bei Jing 100050, China)
Abstract Objective: A kind of detective method for host cells residual DNA of species non-specificity is established by using DOP-PCR. Methods: DOP-primers and conditions of Q-PCR were optimized, so DOP-PCR was established, in order to detective residual DNA of three kinds of host cells (CHO, SP2/0, NS0). Result: the optimized DOP-primer was 5`Tag-N6-ATGTGG 3`. the optimized Q-PCR condition was that the anneal temperature of low stringency cycle rise from 32℃ to 72% by the slope of 3%, and that fluorescence detective conditions of high stringency cycle was 77℃, 30s. the values of R2 of amplification curves of three cells were above 0.99. the recoveries of them were between 80% and 132%, whose RSDs were below 25%. the recoveries of DOP-PCR and general Q-PCR were between 80% and 130%, whose RSDs were below 20%.Conclusion: A kind of detective method for host cells residual DNA of species non-specificity is established by using DOP-PCR, it provides a new idea of detecting host cell residual DNA of monoclonal antibodies.
Key words: DOP, Q-PCR, Residual DNA, Monoclonal antibodies
单抗药物的质量控制中残余DNA一直是人们关注的热点。由于单抗药物通常使用连续传代细胞(如CHO,SP2/0及NS0细胞等)进行工业化生产,而该类细胞是由于调控生长的基因失调, 使得传代细胞系具有无限的寿命。因此, 理论上认为这类连续传代细胞系的DNA具有使其他细胞生长失控和产生致瘤活性的潜在能力, 需要对其DNA残留量进行质量控制[1-2]。在国际上,世界卫生组织(WHO)也发布了大量有关残余DNA的技术文献[2-5],从而指导残余DNA的质量控制。
目前关于外源性DNA残留检测,各国药典收录的附录方法不尽相同:《中国药典》(ChP)收录了地高辛杂交法和荧光染色法,《美国药典》(USP)收录了定量PCR法, 而《欧洲药典》(EP)只收录了DNA杂交法,具体见表1。DOP-PCR,又称简并引物PCR,是引物为5`Tag-Nx-XXXXXX 3`(Nx为x个随机碱基,XXXXXX为6个固定碱基,该引物又称随机引物)的定量PCR。此类PCR先利用随机引物进行5轮非严谨性扩增,借以增加模板的量,所得产物随后进行40轮严谨性扩增,并进行荧光定量,检测模板DNA浓度。它的特点是具有物种非特异性,无需针对某物种的富含DNA重复序列设计特异性引物,并且是对全基因组扩增而非富含序列的扩增。该方法常用于全基因组测序,也可用于未知病毒检测和法医学痕量分析[6-8]。本文通过建立并优化物种非特异性的DOP-PCR法来进行单抗药物宿主细胞残余DNA检测。
表1 各国药典残余DNA检测方法的收录情况
|
药典 方 法 |
中国药典 |
美国药典 |
欧洲药典 |
|
地高辛杂交法 |
收录 |
无 |
收录 |
|
荧光染色法 |
收录 |
无 |
无 |
|
定量PCR法 |
无 |
收录 |
无 |
1 材料与方法
1.1 样品
CHO细胞DNA,SP2/0细胞DNA,NS0细胞DNA,均为本室保存。
1.2 主要仪器及试剂
MicroAmp™ Fast Optical 96孔光学反应板(Applied Biosystems,4346906);MicroAmp™光学封板膜(Applied Biosystems,4311971);resDNASEQTM Quantitative CHO DNA Kit(Applied Biosystems,4402085);resDNASEQTM Quantitative NS0 DNA Kit(Applied Biosystems,);SYBR Premix Ex TaqTMII试剂盒(TaKaRa公司);不含核酸酶的水(Cambrex W50-640);Qiagen Genomic DNA tip 500/G(Qiagen,10262);基因组DNA缓冲液试剂盒(Qiagen 19060);蛋白酶K储备液(Qiagen ,19131);20×Tris-EDTA(TE),(Invitrogen, T11493);磷酸盐缓冲盐水(PBS)(GSK ,7101)。
荧光定量PCR仪,Applied Biosystems,7500Fast;NanoDrop2000超微量分光光度计,Thermo,ND2000;涡旋振荡器,Scientific Industries,型号:Vortex-Genie 2;QIAGEN®离心机,Sigma 4-15C;PCR洁净工作台。
1.3 方法
1.3.1 DNA标准溶液的制备
使用不含核酸酶的水稀释,制备CHO、NS0、SP2/0细胞的 DNA系列溶液,1000 pg/μL、100 pg/μL、10pg/μL、1pg/μL、100 fg/μL、10 fg/μL、1 fg/μL的标准品溶液。
1.3.2 DOP引物序列
DOP(5`-CCGACTCGAGNNNNNNATGTGG-3`)[7]使用不含核酸酶的水溶解引物干粉,工作引物浓度10μM。
1.3.3 PCR反应体系
1.3.3.1 DOP-SYBR PCR扩增反应体系(20 μL):
按下表2进行反应体系的配制,各样品制备3个复孔,加样过程先将除样品DNA以外的各组分预混合,15μL/孔,最后加入标准品/待测样品DNA 5μL/孔,加好后盖紧盖子,将PCR96孔板于台式离心机中瞬时离心,使混合液集中于底部。
表2 DOP-SYBR PCR扩增反应体系
|
组成成分 |
体积(μL) |
|
SYBR(2×) |
10 |
|
Dop-引物(10μM) |
2 |
|
ROX Ⅱ |
0.4 |
|
Rnase free water |
2.6 |
|
标准品/待测样品 |
5 |
1.3.3.2 Taqman PCR扩增反应体系(20 μl):
按下表3进行反应体系的配制,各样品制备3个复孔,加样过程先将除样品DNA以外的各组分预混合,15μL/孔,最后加入标准品/待测样品DNA 5μL/孔,加好后盖紧盖子,将PCR 96孔板于台式离心机中瞬时离心,使混合液集中于底部。
表3 Taqman PCR扩增反应体系
|
组成成分 |
体积(μL) |
|
10×CHO DNA Real-Time PCR Assay Mix |
2 |
|
2×Environmental Master Mix |
10 |
|
Negative Control |
3 |
|
标准品/待测样品 |
5 |
1.3.4 PCR反应程序
1.3.4.1 DOP-SYBR PCR反应程序
Stage1:95℃预变性5 min;
Stage2:95℃变性1min,32℃退火1 min 30s,72℃延伸3min,5个循环(以3%的升温速率由32℃升温至72℃);
Stage3:95℃变性15s,62℃退火1 min,72℃延伸1min,77℃(采集荧光信号)1min,45个循环;
Stage4:溶解曲线:95℃,15s;60℃1 min;95℃变性15s,60℃,15s。
1.3.4.2 Taqman PCR反应程序
Stage1:95℃预变性5 min;
Stage2:95℃变性15秒,60℃退火(延伸)1 min 40个循环。
1.3.5 回收率及精密度
回收率及精密度测定的DNA样品为TE buffer加入1pg或0.01pg的DNA,按照1.3.4项方法进行定量PCR 扩增,平行做3孔。应用7500 FAST 软件分析数据,绘制标准曲线,计算回收DNAD的初始浓度,并按下式计算DNA 回收率,验证方法的准确性。试验重复3 次,计算相对标准偏差(RSD),验证方法的精密性。
DNA 回收率(%)=样品测定值÷DNA 标准品理论值× 100%
2 结果
2.1 DOP-PCR方法优化
在文献参数[7]的基础上,优化低严谨扩增阶段32℃升温至72℃的斜率和读数温度:分别选择斜率100%、5%、3%及2%,最终确定斜率为3%;检测温度分别选择72,76,77,78℃,最终确定为77℃。优化后的熔解曲线如图1,R2达到0.99,NTC的Ct值为40.77±0.40,接近1fg/μL浓度的Ct值,故定量限为10 fg/μL。
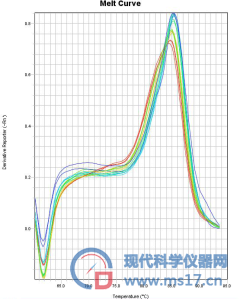
图1 DOP-PCR优化条件后的熔解曲线
2.2 DOP-PCR扩增结果
定量PCR 扩增曲线光滑平稳,峰值较高,且指数增长期、线性增长期及平台期明显,符合定量检测要求,见图2,图3和图4。DNA含量在1×10-2~104 pg/μL 范围内曲线线性良好,表明本方法检测CHO、SP2/0及NS0细胞的DNA残留最低准确定量浓度可达1×10-2 pg/μL。
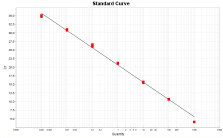
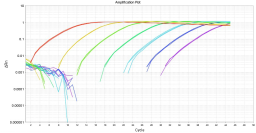
图2a CHO细胞DNA标准曲线 图2b CHO细胞DNA扩增曲线
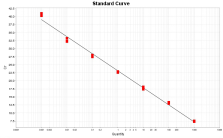
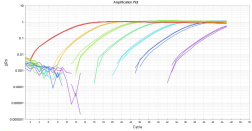
图3a SP2/0细胞DNA标准曲线 图3b SP2/0细胞DNA扩增曲线
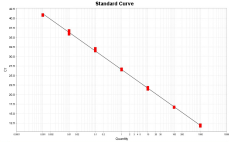
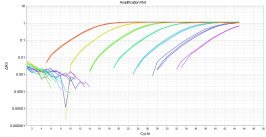
图4a NS0细胞DNA标准曲线 图4b NS0细胞DNA扩增曲线
2.3 DOP-PCR的方法学验证
精密度和准确性 试验结果显示,DNA加标量为1pg时,不同细胞的DNA回收率相近,均值均在80%~130%之间,3次测定的RSD 均小于25%,见表4,表明DOP-PCR法准确性和精密性符合要求。
表 4 不同细胞的DNA回收率
|
细胞 |
1pg加标的DNA回收率(%) |
均值(%) |
SD(%) |
RSD(%) |
||
|
第1次 |
第2次 |
第3次 |
||||
|
CHO |
92.9305 |
76.7532 |
73.2542 |
80.98 |
10.50 |
12.97 |
|
SP2/0 |
69.7534 |
95.4954 |
104.808 |
90.02 |
18.16 |
20.17 |
|
NS0 |
107.398 |
129.75 |
132.417 |
123.19 |
13.74 |
11.15 |
2.4 DOP-PCR与传统定量PCR比较
由于单抗制品主要是由CHO细胞表达,故比较两种方法采用CHO细胞DNA进行试验。结果表明DOP-PCR与传统定量PCR的DNA回收率的均值均在80%~ 130%之间,3次测定的RSD 均小于20%,见表5和6。传统定量PCR见图5。
表5 DOP-PCR与传统定量PCR的DNA回收率比较(1pg加标)
|
方法 |
1pg加标的DNA回收率(%) |
均值(%) |
SD(%) |
RSD(%) |
||
|
第1次 |
第2次 |
第3次 |
||||
|
DOP-PCR |
92.9305 |
76.7532 |
73.2542 |
80.98 |
10.50 |
12.97 |
|
定量PCR |
88.2 |
93.5 |
106.4 |
96.03 |
9.36 |
9.75 |
表6 DOP-PCR与传统定量PCR的DNA回收率比较(0.01pg加标)
|
方法 |
0.01pg加标的DNA回收率(%) |
均值(%) |
SD(%) |
RSD(%) |
||
|
第1次 |
第2次 |
第3次 |
||||
|
DOP-PCR |
85.3185 |
104.559 |
110.301 |
100.06 |
13.08 |
13.07 |
|
定量PCR |
110 |
89 |
115 |
104.67 |
13.80 |
13.18 |
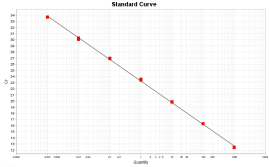
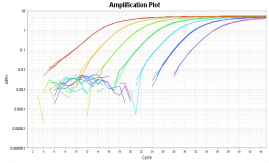
图5a CHO细胞定量PCR标准曲线 图5b CHO细胞定量PCR扩增曲线
3 讨论
不同生物制品应该根据DNA残留量标准选择最优的方法。地高辛杂交法的优势在于成本低廉,无需仪器检测,基因组覆盖率较高,缺点是实验操作周期长、干扰因素多、重复性差,不能进行定量分析,只能限量实验,且灵敏度低,对于单抗制品的实验难以达到检测需要;荧光染色法与杂交法相比较,具有简便、快速、自动化程度高、可定量分析,缺点是定量限低(1.25ng/mL),更适用于大肠杆菌等表达体系,及残余DNA限度标准较低(10ng/剂量)、人用剂量较小的生物制品,而针对毫克级的单抗制品缺乏适用性;定量PCR法与DNA杂交法和荧光染色法相比,该方法具有更高的检测灵敏度,定量限可达1fg,DNA含量在1fg~10ng范围内线性良好,可以满足剂量较大而超出杂交法或荧光染料法检测范围的单抗制品的质控要求,缺点是有种属特异性(CHO,SP2/0,NS0细胞等),并且引物是针对某物种的富含DNA重复序列设计的,可能存在部分非富含DNA序列漏检的风险,具体见表7。比如对于大肠杆菌表达的细胞因子,残余DNA限度为不超过10ng/剂量,目前杂交法就能够满足检测需求;荧光染色法对于重组人EPO(CHO细胞)残余DNA的检测较为适用;对于单抗则更适用定量PCR进行检测[1]。单抗制品的人用剂量一般毫克级,且主要由CHO细胞表达,还有一部分是SP2/0细胞和NS0细胞表达,其宿主DNA残留量一般要求不高于100pg/剂量[9-11]。
表7 三种残余DNA检测方法比较
|
方法 |
地高辛杂交法 |
荧光染色法 |
定量PCR法 |
|
优点 |
成本低廉 无需仪器检测基因组覆盖率较高 |
定量法 简便、快速 自动化程度高 |
定量法高(1fg/mL) 检测灵敏度 1fg~10ng范围内线性良好 |
|
缺点 |
周期长 限量法 灵敏度低 |
定量限低 (1.25ng/mL) |
种属特异性引物 针对富含序列检测 非富含序列漏检风险 |
DOP-PCR是一类特殊的定量PCR,其引物称为DOP引物,序列为5`Tag-Nx-XXXXXX 3`(Nx为x个随机碱基,XXXXXX为6个固定碱基),其中Nx的碱基序列可能为4x种可能性,故又称随机引物。这些引物随机结合到DNA模板上,进行5轮非严谨性扩增(32℃退火,斜率为3%,缓慢升温到72℃进行延伸),见图5,所得产物随后进行40轮严谨性扩增(62℃退火),最终在77℃,30s进行荧光定量,检测模板DNA浓度。在DOP-PCR方法优化过程中,尝试了非严谨性扩增、读数条件等多个参数的优化,最终发现非严谨性扩增的条件设定为32℃退火,斜率为3%缓慢升温到72℃进行延伸,读数条件为77℃,30s,在该条件下,三个物种细胞10fg的Ct值与无关对照(NTC)差异最大,选为最优条件。1 fg/μL的Ct值与无关对照(NTC)无差异,无法进行有效检测。
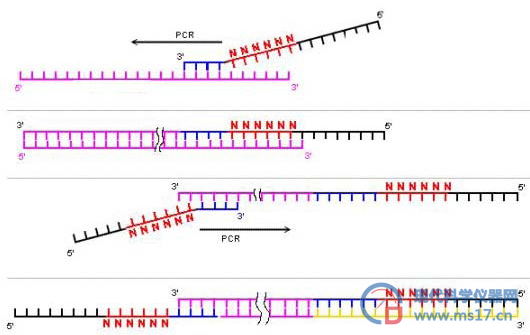
图6 DOP-PCR的扩增原理
DOP-PCR与传统定量PCR相比,无需根据种属的富含DNA序列设计特异性引物,随机引物适用于包括CHO,SP2/0和NS0细胞等在内的表达体系;传统定量PCR的定量限为1fg/μL,由于DOP引物为非特异扩增,可能形成引物二聚体,导致NTC的本底Ct值较大,接近10fg/μL的Ct值,无法与1fg/μL有效区分,故DOP-PCR的定量限只能达到为10fg/μL,而这对于单抗制品残余DNA pg级的标准规定,二者均是适用。
本研究基于CHO细胞基因组DNA进行方法摸索和优化,但尚需实际单抗制品进一步开展全面的方法学验证,从而评价该方法在单抗制品宿主细胞残余DNA检测中的可行性。本研究引入了非物种特异性DOP-PCR法,为单抗制品宿主细胞残余DNA检测提供了新思路和未来发展的应用前景。
参考文献
[1] 王兰,王军志. 关于生物制品残余DNA质量控制问题. 中国新药杂志,2011,20(8):678-683.
[2] WHO Expert Committee on Biological Standardization. Forty-seventhReport.
Requirements for the use of animal cells as in vitro Substrates for the production of biological [R]. Geneva: World Health Organization, 1998 ( WHO Technical Report Series, No.878, annex 1 ).
[3] WHO Study Group. Acceptability of cell substrates for production of biological [R] .1987 ( WHO Technical Report Series, No747 ) .
[4] WHO Expert Committee on Biological Standardization. Thirtysixth Report. Requirements for continuous cell lines used for biological production [R] . Geneva: World Health Organization, 1987, Annex3 (WHO Technical Report Series, No.745)
[5] WHO Expert committee on biological standardization. Recommendations for the evaluation of animal cell cultures as substrates for the manufacture of biological medicinal products and for the characterization of cell banks[R]. Geneva: World Health Organization, 18-22 October 2010.
[6] Telenius H, Carter NP, Bebb CE, et al. Degenerate Oligonucleotide-Primed PCR: General amplification of target DNA by a single degenerate primer[J]. Genomics. 1992, 13: 718-725.
[7] Min-Jung Kang, Hannah Yu, Sook-Kyung Kim, et al. Quantification of Trace-Level DNA by Real-Time Whole Genome Amplification [J]. PLoS ONE. 2011, 6 (12): e28661.
[8] 吴小兵,任鲁风,侯云德等. 用SARS冠状病毒全基因组芯片杂交方法分析SARS-CoV. 中国生物工程杂志,2003,23(7):89-93.
[9] 王兰,李萌,高凯等. 磁珠分离结合定量PCR 法检测单克隆抗体产品中CHO 细胞DNA残留量. 中国生物制品学杂志,2014,24(7):555-558.
[10] Lan Wang, Chunming Rao, Kai Gao, et al. Development of a reference standard of Escherichia coli DNA for residual DNA determination in China [J]. PLoS ONE. 2013, 8 (9) : e74166.
[11] 王军志.生物技术药物研究开发和质量控制[M] .第二版. 北京:科学出版社,2007:98.
(资讯来源:《现代科学仪器》,由“现代科学仪器网”官方发布,转载请注明来源)

关注本网官方微信 随时订阅权威资讯

